国立研究開発法人 産業技術総合研究所【理事長 中鉢 良治】(以下「産総研」という)人工知能研究センター【研究センター長 辻井 潤一】オーミクス情報研究チーム 兼 創薬基盤研究部門【研究部門長 亀山 仁彦】付 兼 産総研・早大生体システムビッグデータ解析オープンイノベーションラボラトリ【ラボ長 竹山 春子】(以下「CBBD-OIL」という)付 齋藤 裕 研究員、オーミクス情報研究チーム 兼 創薬基盤研究部門付 亀田 倫史 主任研究員、生物プロセス研究部門【研究部門長 鈴木 馨】応用分子微生物学研究グループ 兼CBBD-OIL付 北川 航 主任研究員、生命工学領域【領域長 松岡 克典】 田村 具博 研究戦略部長 兼 CBBD-OIL付らは、微生物を用いたタンパク質の生産量向上のために導入する遺伝子配列を、情報技術に基づいて設計する手法を開発した。
微生物を用いた物質生産では、対象の微生物に異種由来の遺伝子を人工的に導入して、その微生物が本来持たないタンパク質を生産させる。その際、目的タンパク質の生産量を向上させるために、導入する遺伝子のDNA配列を適切に設計する行程(コドン最適化)が重要となる。従来のコドン最適化の研究は大腸菌などの実験が行いやすい研究用の微生物が主な対象で、放線菌などのバイオ産業の物質生産の現場で用いられる微生物については確立されたコドン最適化手法がなかった。今回、産総研の持つ大規模なタンパク質生産実験データから解析によってルールを抽出して新しいコドン最適化手法を開発し、ロドコッカス属放線菌で手法の有効性を実証した。この手法で設計された遺伝子配列は元の配列の先頭部分にしか変異を含まないため、安価な実験コストで合成できる。今回開発した技術により、医療・食品・環境などさまざまな分野でのバイオものづくりの加速が期待される。
なお、この成果は、2019年6月6日(英国夏時間)に論文誌Scientific Reportsに掲載される。
 |
| 情報技術による遺伝子配列設計でタンパク質生産量を向上 |
近年、微生物を用いた物質生産が注目され、医薬品や機能性食品の原料、バイオ燃料の生産などさまざまな応用例がある。微生物に所望の物質を生産させる方法には、対象となる微生物に異なる生物種由来の遺伝子を人工的に導入して、その微生物が本来持たないタンパク質を生産させる方法がある。その際、目的タンパク質の生産量向上のためには、導入する遺伝子のDNA配列を設計する行程が重要となる。この配列設計は、タンパク質のアミノ酸配列が変わらないようにDNA配列でのコドンの使用パターンを最適化する設計であるため、「コドン最適化」と呼ばれている。
従来のコドン最適化では、対象の微生物の内在性遺伝子で高頻度で使われているコドンを、導入する遺伝子でも使用する方法などが有効と信じられてきたが、本当に生産量向上に有効であるかを大規模な実験データに基づいて詳細に検証する研究は、あまり行われていない。最近、大腸菌などについては、そうした研究が少しずつ行われはじめたが、その知見が他の微生物にも適用できるかは未知であり、バイオ産業の物質生産の現場で用いられるさまざまな微生物の多くには、確立されたコドン最適化手法がなかった。
放線菌の一種、ロドコッカス・エリスロポリス(Rhodococcus erythropolis)は、4~35 ℃の幅広い温度域で増殖でき、有機溶媒耐性や脂肪族・芳香族・複素環式化合物を変換する生体触媒活性など、数多くの特長を持つ微生物である。大腸菌などの汎用宿主とは大きく異なる性質から、次世代宿主の候補として世界的に期待されている。しかし、ロドコッカス・エリスロポリスのコドン最適化手法は確立されておらず、この微生物を応用する上での障害となっていた。
産総研は、ロドコッカス・エリスロポリスを物質生産に利用するための研究を継続的に行い、その有用性と安全性を証明してきた(産総研主な研究成果 2014年7月1日)。特に、タンパク質生産実験を多数の遺伝子について行っており、ロドコッカス・エリスロポリスでは世界最大規模のデータを持っている。また、大規模データの解析によって有用な知見を抽出するための研究を行ってきた。今回、これらの産総研の強みを融合させて、ロドコッカス・エリスロポリスにも適用できる新しいコドン最適化手法の開発に取り組んだ。
なお、今回の本研究開発は、国立研究開発法人 新エネルギー・産業技術総合開発機構(NEDO)の委託事業「植物等の生物を用いた高機能品生産技術の開発」による支援を受けて行った。
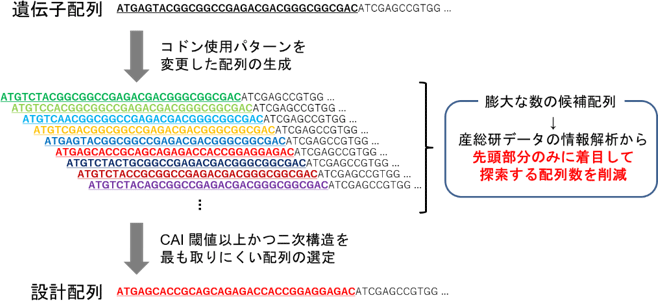
産総研は、ロドコッカス・エリスロポリスL-88株に別種の放線菌ストレプトマイセス・セリカラー(Streptomyces coelicolor A3(2)株)由来の遺伝子を導入したタンパク質生産実験データを保有している。このデータは、204個の遺伝子の遺伝子配列とタンパク質生産量とのデータであり、これらの解析を行った。遺伝子配列からさまざまな配列特徴量の計算を行い、タンパク質生産量と配列特徴量の相関を評価した結果、遺伝子配列の先頭部分のmRNAの二次構造形成度とCAI(Codon Adaptation Index)という配列特徴量が、タンパク質生産量と高い相関を示した。この結果に基づき、二次構造を取りにくく、またCAIが高くなるように遺伝子配列の先端部分だけを改変するコドン最適化手法を開発した(図1)。この手法では以下の手順でタンパク質生産量を増加させるために最適と考えられる配列を作成する。
(1)元となる遺伝子配列に対して、生産されるタンパク質の先頭11個のアミノ酸配列が同一のまま、コドンの使用パターンだけが変更された遺伝子配列(mRNA配列)全てをコンピューター上で作成する。
(2)それぞれの遺伝子配列の先頭部分について転写開始点から11コドン目までの二次構造形成度と、11個のコドンのCAIを計算する。
(3)全配列のうち、CAIが指定された閾値以上に高い配列から、二次構造を最も取りにくい配列を探索し、最適配列の候補とする。
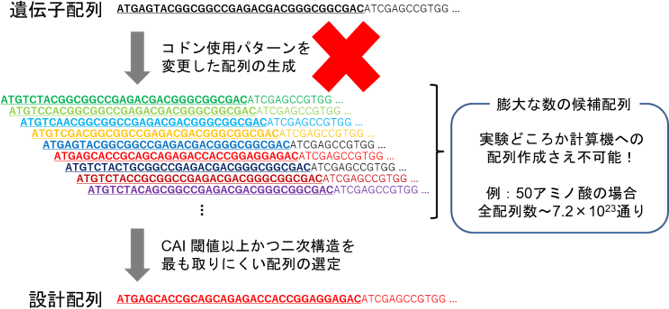
今回、産総研が保有する実験データの解析によって、タンパク質生産には遺伝子配列の先頭部分が重要と分かったことは意義深い。一般に、1個のアミノ酸は平均して3個のコドンを持つので、例えばアミノ酸50残基から構成される小さなタンパク質についてコドン使用パターンを変えた全遺伝子配列は、350≒7.2×1023通り(およそ1億×1億×1億×1億)という膨大な数となりコンピューター上に配列を作成することさえ不可能である(図2)。今回の成果により、コドン使用パターンを変更する範囲を遺伝子配列の先頭部分、約10コドンに絞り込めたため、コドン使用パターンを変えた全配列数は、310≒59,000通りと大幅に削減できて、計算可能となったため、実行可能な今回の手法を開発することができた(図1)。
 |
| 図1 今回開発した手法によるコドン最適化の概略 |
 |
| 図2 遺伝子配列の先頭部分ではなく、全体に対してコドン最適化を行った場合 |
今回開発したコドン最適化法の有効性を検証するために、12個の遺伝子(配列長100~400アミノ酸)について最適配列を設計し、ロドコッカス・エリスロポリスに導入してタンパク質生産量を評価した。その結果、9個の遺伝子(75 %)で野生型配列よりも生産量が向上した(表1)。特に、野生型配列によるタンパク質生産量が少なかった5個の遺伝子については、全てが今回の手法によって設計した配列によって生産量が向上した(図3;TetR転写因子の例)。この結果は、本手法が生産の難しいタンパク質に対して特に有効であることを示している。また、今回の手法はタンパク質生産量の向上だけでなく、生産量低下にも有効であった。なお、これらの検証では、12個の遺伝子それぞれについて10種類の遺伝子配列によるタンパク質生産実験を3回ずつ実施した(12遺伝子×10配列×3回=合計360実験)。これはこれまでのコドン最適化の研究に比べて非常に大規模な実験であり、それによって検証された今回の手法の信頼性は高いと考えられる。
 |
| 表1 今回の手法で設計した遺伝子配列によるタンパク質生産量向上の有効性検証結果 |
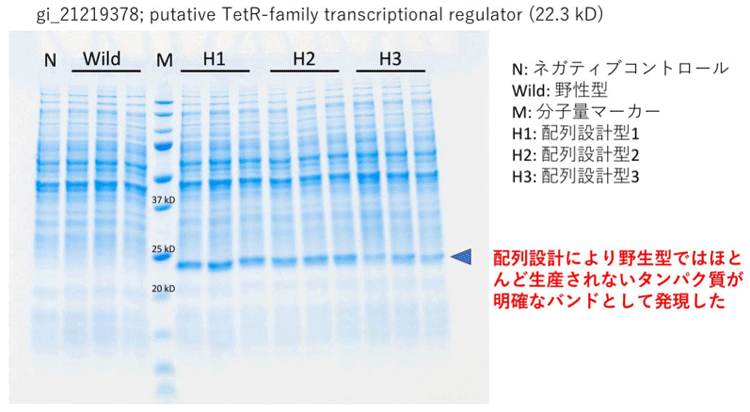 |
| 図3 配列設計による発現量の改善例 (タンパク質電気泳動による発現解析) |
今回の手法は、遺伝子配列全体でなく先頭部分だけを改変するため、設計した配列を合成する際に、全長遺伝子合成のようなコストの高い方法ではなく、変異導入プライマーによるPCRという、簡便で安価な方法で合成できるという利点がある。また、CAIを対象の微生物に合わせることで、大腸菌などロドコッカス属以外のさまざまな微生物におけるタンパク質生産にも応用できる可能性がある。
今回開発した技術をさまざまな微生物によるタンパク質生産に適用して有効性を実証するとともに、物質生産の効率向上に役立てていく。また、タンパク質生産を低下させることできるため、物質生産の精密な制御を行うための技術として発展させていく。