国立研究開発法人 産業技術総合研究所【理事長 中鉢 良治】(以下「産総研」という)生物プロセス研究部門【研究部門長 田村 具博】合成生物工学研究グループ 宮崎 健太郎 研究グループ長は、北海道大学【総長 名和 豊春】大学院理学研究院 北原 圭 特任助教らの協力を得て、環境バクテリアの16SリボソーマルRNA(16S rRNA)遺伝子ライブラリーの中から、淋菌感染症治療に用いられる抗生物質のスペクチノマイシンに対して耐性を示す16S rRNA遺伝子を複数発見した。さらに、耐性遺伝子の変異解析の結果、新たな耐性変異を発見した。
rRNAは抗生物質の主要なターゲットの一つであり、rRNAに耐性変異が生じると、抗生物質が効かなくなる。このため、耐性変異の情報は耐性菌の早期発見に有用であるが、これまでrRNAの耐性変異の同定は困難で、耐性変異に関する情報は十分に蓄積されていなかった。今回、大腸菌を宿主として異種バクテリア由来の16S rRNAを機能解析する産総研独自の技術が、抗生物質耐性の検証に適用できることが分かった。この技術により、環境中のバクテリア由来の16S rRNA遺伝子から4種の耐性遺伝子を同定し、それらの遺伝子の変異解析により新たにスペクチノマイシンに対する3つの耐性変異を発見した。今回の方法をさまざまな抗生物質の耐性変異の検証に適用することで、耐性菌の発見・診断に有用な、耐性変異のデータベース構築が期待される。
なお、この研究の詳細は、2018年4月3日(英国時間)に英国科学雑誌Scientific Reportsにオンライン掲載される。
 |
|
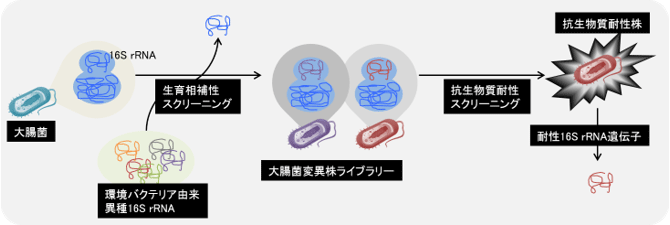
16S rRNAの機能解析技術を用いた抗生物質耐性スクリーニングの概要 |
抗生物質は病原性菌を死滅させる効果を持つため、感染症を抑制するため抗生物質の開発が行われている。しかし、多くの場合、病原性菌は他の生物と同様に抗生物質の攻撃を逃れる仕組みを持ち、抗生物質耐性を獲得する。耐性菌の早期発見・診断には耐性変異に関する情報が重要であるが、十分な知見が蓄積されていない。その原因の一つは、多くの抗生物質の標的であるrRNAの機能解析法が確立されていないことである。rRNAは生体内で最も多く生産される重要な遺伝子であるため、多くのバクテリアはゲノム上に複数のコピーを持っている。そのため、単一のバクテリア内にも、耐性を獲得した遺伝子とそうでない遺伝子が混在することがある。複数のコピーの一部の遺伝子だけが耐性を獲得した場合、一般的には強い耐性を示さず、耐性変異が「潜伏」した状態となっている。こうした潜伏状態の耐性変異を確実に検出するには、ゲノム全体に含まれる複数のrRNAについて、個別に抗生物質耐性の有無を解析する必要があるが、これまで、解析手法がなく、その開発が求められていた。
これまで産総研では、リボソームの機能解析手法の一つとして16S rRNAを生物種間で交換する遺伝学的手法を開発し、翻訳装置であるリボソームに翻訳以外の機能(RNA分解酵素阻害活性)を発見するなど、リボソームの機能解析に革新をもたらした(2011年11月23日 産総研プレス発表)。また、大腸菌とは進化系統分類の「綱」や、さらに上位の「門」のレベルで異なる生物種の16S rRNAが大腸菌遺伝子と機能的に互換であることを発見した(2012年10月30日、2017年8月30日 産総研プレス発表)。
今回、この16S rRNAを生物種間で交換する手法を用いて、環境中のさまざまなバクテリア由来の16S rRNAの中から、16S rRNAに作用する抗生物質の一つであるスペクチノマイシンに対して耐性を示す16S rRNA遺伝子をスクリーニングし、発見した耐性16S rRNA遺伝子の変異解析による新たな耐性変異の同定に取り組んだ。
なお、この研究は、独立行政法人 日本学術振興会の科学研究費助成事業 挑戦的萌芽研究「リボソーマルRNAの抗生物質耐性変異の網羅的解析」(平成26~27年度)による支援を受けて行った。
16S rRNA遺伝子は生育に必須の遺伝子で、これを欠いた大腸菌は生育できない。また、16S rRNA遺伝子は生物種に固有の遺伝子であり、生物種間では機能の互換性がないと考えられてきた。しかし、産総研では2012年に、大腸菌の16S rRNA遺伝子欠損株に異種バクテリア由来の16S rRNAを組み込んで、生育できるかどうかを調べ(生育相補性スクリーニング)、多くの異種バクテリア由来の16S rRNAにより大腸菌16S rRNA遺伝子欠損株が生育できることを見出した。
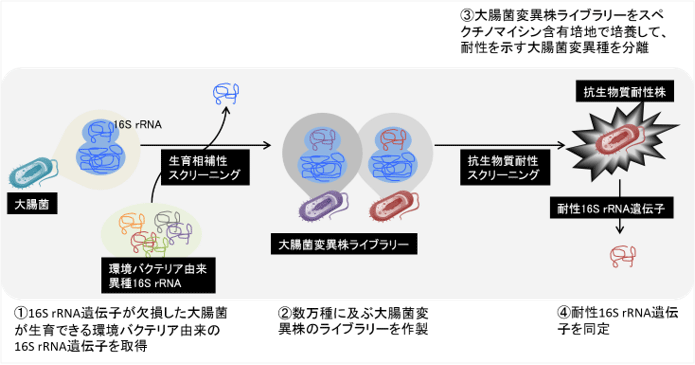
そこで、今回この方法を利用して、環境中のバクテリアの16S rRNAの抗生物質耐性の有無をスクリーニングするため、まず、大腸菌16S rRNA遺伝子欠損株が生育できる環境バクテリア由来の16S rRNA遺伝子を大量に取得し、数万種の大腸菌変異株からなるライブラリーを作製した。次に、スペクチノマイシンに対して耐性を示す16S rRNA遺伝子を得るため、この大腸菌変異株ライブラリーを、スペクチノマイシンを含む培地で培養し、生育できた大腸菌変異種、すなわち耐性を示す大腸菌変異種を分離した。これらの16S rRNA遺伝子の解析により、4種のスペクチノマイシン耐性16S rRNA遺伝子を同定した(図1)。
 |
|
図1 抗生物質耐性スクリーニングの流れ |
これら4種のスペクチノマイシン耐性16S rRNA遺伝子の配列を決定し、米国バイオテクノロジー情報センター(NCBI)の遺伝子配列データベースから、耐性16S rRNA遺伝子に類似した遺伝子を検索した。データベース中の相同遺伝子はスペクチノマイシン耐性ではないと考えられるため、耐性遺伝子とデータベース中の遺伝子の配列を比較すれば、耐性遺伝子中の耐性変異を推定できる(図2)。例えば、環境由来のスペクチノマイシン耐性遺伝子の一つであるmgSpc2では1066番目の塩基がTであるのに対し、NCBIの遺伝子配列データベースに見出された相同遺伝子(図2ではmgSpc2homと表記)ではCである。この場合、1066番目の塩基CがTに置き換わることで耐性になったと推定できる。
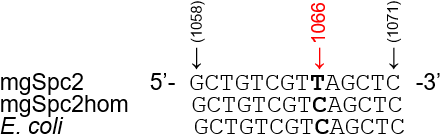 |
図2 16S rRNA遺伝子(一部の領域)の塩基配列の比較
(上段)スペクチノマイシン耐性を示す4種の16S rRNA遺伝子の一つ
(中段)NCBIの遺伝子配列データベースから検索した相同遺伝子
(下段)大腸菌の16S rRNA遺伝子 |
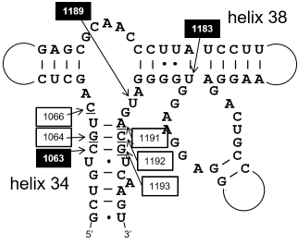
この耐性変異の推定が正しいかどうかを検証するため、環境中のバクテリア由来のスペクチノマイシン耐性16S rRNA遺伝子の耐性変異を、部位特異的変異導入法により相同遺伝子に含まれるタイプの塩基に変換して(図2の例ではmgSpc2の1066番目のTをCに変換する)、その大腸菌変異種が耐性を失うかどうかを調べた。また、推定された耐性変異をスペクチノマイシン非耐性大腸菌の16S rRNA遺伝子に組み込んで耐性が現れるかどうかを確認した。これらの検証の結果、既知の5つの耐性変異のほかに、新たに3つの変異を同定した(図3)。
スペクチノマイシンはバクテリアリボソームに作用する抗生物質として50年以上前に開発され、臨床応用歴も長く、その耐性機構や耐性変異についても多くの研究が行われてきた。スペクチノマイシンに関する知見は相当に蓄積されてきたにもかかわらず、今回、新しい耐性変異が同定できたことは、環境中には数多くの未知の耐性変異が眠っていることを示唆する。
 |
|
図3 大腸菌16S rRNAの二次構造
スペクチノマイシン耐性変異が集中する領域の拡大図。既知の耐性変異は白地に黒数字、今回見つかった新規耐性変異は黒地に白数字で示した。
|
これまで、バクテリアの複数のrRNAを個別に調べて抗生物質耐性を持つものを特定できる有効な手段はなかったが、大腸菌を宿主として16S rRNAの機能を解析する産総研独自の手法であれば、個々の16S rRNAの機能を調べて抗生物質耐性を持つものを特定できることが分かった。また、この手法はスペクチノマイシン以外の抗生物質にも適用できる。そのため、今後、この手法でさまざまな抗生物質の耐性スクリーニングを行い耐性変異に関するデータを蓄積すれば、耐性菌の早期発見・診断に有用な、抗生物質耐性変異のデータベースの構築も可能である。
今後は、16S rRNAを攻撃する他の抗生物質や、さらに、23S rRNAを攻撃する抗生物質にもスクリーニング範囲を拡張する。それにより、rRNAの変異に基づく抗生物質耐性変異の網羅的なデータベースを構築し、病原菌に対して特定の抗生物質が有効かどうかを迅速に高精度で調べる技術の確立を目指す。