国立研究開発法人 産業技術総合研究所【理事長 中鉢 良治】(以下「産総研」という)生物プロセス研究部門【研究部門長 田村 具博】合成生物工学研究グループ 宮崎 健太郎 研究グループ長(東京大学大学院新領域創成科学研究科 客員教授)は、佃 美雪 元産総研技術研修員(兼)元東京大学大学院新領域創成科学研究科博士課程学生(現:花王株式会社安全性科学研究所)、北海道大学【総長 名和 豊春】大学院理学研究院 北原 圭 特任助教らの協力を得て、バクテリア系統分類の最上位である門レベルで相異なる2種のバクテリアのリボソームに含まれる16SリボソーマルRNA(rRNA)の比較機能解析を行い、16S rRNA遺伝子間の配列の違いの大半が機能に大きな影響を及ぼさないことを発見し、16S rRNA遺伝子が中立進化していることを証明した。
この成果は、この遺伝子が進化系統解析に用いられる分子時計の基本的要件である「進化的な中立性」を満たしていることを、世界で初めて実験的に示したものである。解析結果から、リボソームの進化においては生物種に特異的な塩基配列の変異の大半は、リボソームの機能には影響を及ぼさないことが示唆された。一方、この事実は、生物種間での16S rRNAの組み換えが比較的自在であるということを示唆しており、分子時計としての16S rRNA遺伝子の適性を検討する上での重要な知見として活用されることが期待される。
この研究の詳細は、2017年8月30日(英国時間)に英国科学雑誌Scientific Reportsにオンライン掲載される。
 |
リボソームの30Sサブユニットの立体構造(青:16S rRNA、白:21種類のリボソームタンパク質)
RNA を中心にタンパク質が密に相互作用しながら取り囲む極めて複雑な構造をしている。 |
微生物の進化系統関係を明らかにすることは、生命の誕生とその後の進化過程を解明する上で重要な手がかりを与える。しかし、微生物は動植物と異なり化石がほとんど存在しないため、化石によって系統進化の歴史を推定することは非常に難しい。そのため、微生物の系統関係はこれまで、分子の系統関係で議論されてきた。生物の系統進化を正確に反映する「分子マーカー(分子時計)」の要件は、(1)どの生物にも必ず存在すること、(2)生物種に固有であること、(3)機能が生物種によらず同等であること、(4)進化速度(遺伝子が変異する速度)が生物種や生育環境によらず一定であることが挙げられる。しかし、これらをすべて満たす分子は皆無に近い。
rRNAはすべての生物に必ず存在する生体高分子であり、中でも、16SリボソーマルRNA(16S rRNA)遺伝子は、分子時計と見なされており、原核生物の進化系統分類の指標として用いられている。これまで、遺伝子が持つ約1500塩基から構成される配列の類似度に基づいて微生物の系統分類がなされてきた。しかし、16S rRNAが分子時計としての要件、特に(3)および(4)の要件を満たすかどうかは、これまで実験的に検証されていない。
リボソームは生体内で遺伝情報をもとにタンパク質を合成する翻訳装置であり、16S rRNAはその小サブユニットの中心骨格を形成する分子である。産総研では、16S rRNAを生物種間で交換する遺伝学的手法を開発してリボソームが翻訳以外の機能(RNA分解酵素阻害活性)を持つことを発見した(2011年11月23日 産総研プレス発表)。また、大腸菌とは進化系統分類の「綱」のレベルで異なる生物種の16S rRNAが、大腸菌の16S rRNAと機能的に同等であることを明らかにし(2012年10月30日 産総研プレス発表)、生物種に固有という16S rRNAに対する考え方を一変させた。
今回、大腸菌の16S rRNAと機能的に互換である16S rRNAの系統範囲を明らかにするため、環境ゲノム(メタゲノム)由来16S rRNA遺伝子を用いて大規模な機能スクリーニングを行った。なお、この研究開発は、独立行政法人 日本学術振興会の科学研究費助成事業 基盤研究(B)「大腸菌リボソームの可塑性と表現型進化の機構解明」(平成26~28年度)、科学研究費助成事業 挑戦的萌芽研究「rRNAの置換変異によるリボソーム可塑性の研究」(平成24~25年度)、科学研究費助成事業 新学術領域研究「翻訳システム改変による人工細胞創成」(平成24~25年度)による支援を受けて行った。
16S rRNA遺伝子は細胞の生育に不可欠であり、導入した16S rRNAが機能しないと細胞は生育しない。今回の研究では、16S rRNA遺伝子が欠損している大腸菌に、環境ゲノム由来の16S rRNA遺伝子を導入したところ、プロテオバクテリア門に分類される大腸菌の16S rRNA遺伝子を、門レベルで異なるアシドバクテリア門由来の16S rRNA遺伝子(NS11と命名)と入れ替えても大腸菌が生育することが確認された。しかし、NS11を導入した大腸菌変異株では、増殖能の低下、すなわち増殖倍加時間の伸長が観察された(図1)。
NS11と大腸菌16S rRNA遺伝子は、配列全体にわたって334塩基の違いがあるが、増殖能の低下を招いた領域を大まかに探るために、キメラ解析を行った。16S rRNAは4つのドメインから構成されるが、NS11の各ドメインを大腸菌のものと置き換えた結果、一部のドメインを置き換えることで、元の大腸菌の16S rRNAと同等のレベルまで増殖倍加時間が短縮した。さらに、このドメインにおける塩基配列のうち、NS11と大腸菌で比較して機能的に重要であると考えられる4つの塩基を絞り込み、NS11におけるそれら(A1416-U1484)を大腸菌のもの(G1416-C1484)と置換して相補試験を行ったところ、元の大腸菌と同様のレベルまで増殖倍加時間が短縮した。
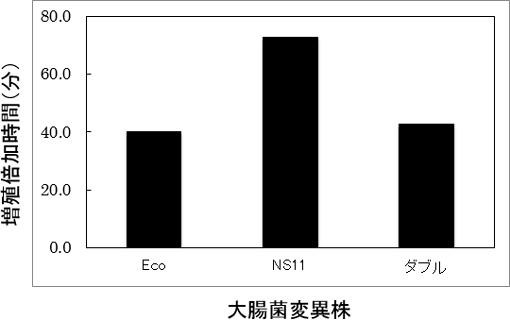 |
| 図1 さまざまな16S rRNAを含む大腸菌の増殖倍加時間 |
| 大腸菌固有の16S rRNA(図中、Ecoと表記)に対して、アシドバクテリア門由来の16S rRNA(図中、NS11)を含む大腸菌変異株の増殖能は低下する(増殖倍加時間の伸長)が、A1416-U1484二塩基(一塩基対)を大腸菌型のG1416-C1484に置換した変異株(図中、ダブル)では増殖能は回復する(増殖倍加時間の短縮)。 |
この結果は、プロテオバクテリア門の大腸菌16S rRNA遺伝子とアシドバクテリア門由来のNS11を分かつ塩基配列の違いの大半(99.4 %)は、生育に影響を及ぼさないことを意味する。すなわち、両生物は共通の祖先型16S rRNAを共有し、それぞれが系統に特異的な変異を蓄積してきたが、それらの大半が機能に影響を及ぼさない「中立変異」であり、16S rRNAが分子時計として必要な要件の一つを持っていることが実験的に初めて証明された。
今回の結果をもとに、リボソームの進化モデルとして「ゆりかごモデル(Cradle model)」を提唱している。このモデルでは、機能に必須なrRNAとタンパク質との相互作用は門レベルでの分化以前に共通祖先の段階で形成され、その後の種分化過程でも引き継がれた。生物種に特異的な変異の大半は機能に無関係である(図2)。このモデルであれば、今回の結果のように、生物種間で16S rRNAの組み換えが比較的自在に行える(水平伝播が起きやすい)こともよく説明できる。一方で、分子時計として16S rRNAが持つとされている別の要件「種固有性」に対しては疑問を呈する結果となった。今後、16S rRNA遺伝子の水平伝播の可能性やその頻度について明らかにする必要がある。
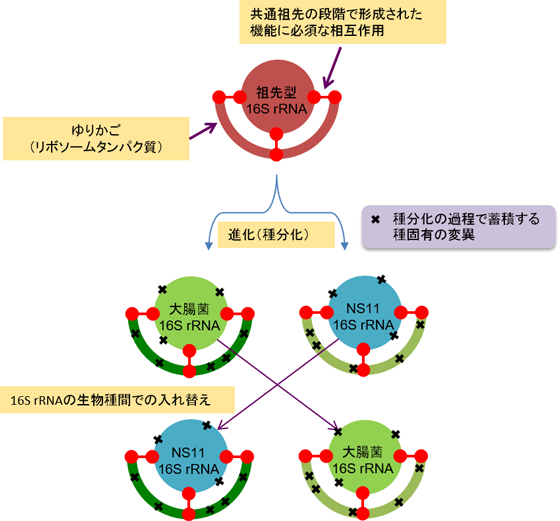 |
| 図2 リボソーム進化のゆりかご(Cradle)モデル |
祖先型リボソームは進化の過程で生物種に特異的なさまざまな変異を蓄積するが、その変異により機能発現に必要な相互作用(図中の  )が破壊されることはない。このため、RNAを生物種間で入れ替えても相互作用は維持され、リボソームがもつ翻訳活性機能も維持される。 )が破壊されることはない。このため、RNAを生物種間で入れ替えても相互作用は維持され、リボソームがもつ翻訳活性機能も維持される。 |
今後は、アシドバクテリア門以外のバクテリアの各門の個別機能解析などにより、今回の結果で示唆されたリボソームの基本的な構造の形成段階が、バクテリア種分化におけるどの段階で生じたのかを検証する予定である。