国立研究開発法人 産業技術総合研究所(以下「産総研」という)バイオメディカル研究部門 関口勇地 総括研究主幹らは株式会社島津製作所(以下「島津製作所」という)と共同で、質量分析技術を利用し、従来不可能であった多様な原核微生物種の迅速同定を可能にする技術を開発しました。
本技術では、原核微生物のゲノム情報から推定した大規模理論タンパク質量データベース構築と質量分析結果の解析アルゴリズム開発により、未培養微生物を含む多様な原核微生物種の迅速同定を実現しました。マトリックス支援レーザー脱離イオン化飛行時間型質量分析法(MALDI-TOF MS)による菌体構成物の質量分析に基づく迅速微生物同定法のための従来にない解析プラットフォームであり、従来の培養菌体を利用した実測質量データベースと比較して、同定可能な微生物種が10倍以上増加しました。また、従来不可能であった未培養微生物の迅速同定も可能になります。本技術は、多様な微生物の迅速同定に役立つため、ヒトや動物などの感染症原因微生物の特定、食品分野の微生物検査、環境微生物分析の高度化、迅速化のみならず、有用微生物のスクリーニングなどの研究開発の高度化、迅速化に貢献します。
なお、この技術の詳細は、2023年12月5日(英国時間)に「Genome Biology」誌に掲載されます。
環境中には多様な原核微生物(バクテリア、アーキア)が存在しており、ヒトや動物などの健康、食品などの品質、環境浄化や地球上の物質循環に極めて密接に関係しています。このため、感染症の臨床診断、食品の衛生管理、環境衛生の評価などにおいて、それら微生物を培養、分離した後、分類学的に同定する検査が実施されています。MALDI-TOF MSに基づいた分離培養微生物の同定技術は、核酸検査などと比較し、最小限の試料前処理および質量分析の迅速性により、極めて短時間で微生物同定が可能な技術です。その迅速性により、主に感染症などの臨床検査の分野においてMALDI-TOF MSは広く利用されています。
一方、食品や環境など、病原細菌以外の多様な微生物が検出される分野において本技術の応用は遅れています。その理由として、MALDI-TOF MS微生物同定には既知微生物の質量スペクトル情報に基づくデータベース(ライブラリ)が必要であることが挙げられます。培養分離株を使用して実験的に決定される実測質量スペクトル情報を参照情報とする従来のライブラリは通常数千程度の微生物種に限定されており、ライブラリに掲載されていない微生物種の同定は不可能です。さらに、地球上に存在する原核微生物種の半分以上は実験室では培養されていない未培養微生物、難培養微生物であり、それら微生物群を捕捉することも不可能でした。
また、複数種類の微生物群で構成される生態系はマイクロバイオームと呼ばれ、その重要性が注目されています。とりわけ、ヒト腸内などのマイクロバイオームと疾患、健康の関係性が明らかにされつつあり、疾患の治療を目的とし有用微生物株を生菌製剤(医薬品)として活用するための研究開発が活発化しています。この分野の研究開発では、疾患の発症や治療に関わる微生物群を培養、分離することが求められますが、そのためには数千から数万の分離株を迅速に同定し、目的微生物をスクリーニングする必要があります。そのようなヒトのマイクロバイオームを「培養」する取り組みを加速するためにも、未培養微生物群を含む広範囲な原核微生物群を迅速に同定する技術が必要でした。
産総研は、マイクロバイオームを構成する微生物の種類と量の計測(マイクロバイオーム解析)とその精度管理技術に関する研究開発を行ってきました(2016年12月14日 産総研プレス発表、2021年4月29日 産総研プレス発表)。また、日本の健康者腸内マイクロバイオームの理解と産業活用を目的に、その構成微生物群の大規模解析、構成微生物群の分離培養、ゲノム解析などを行ってきました。
島津製作所は、田中耕一記念質量分析研究所を中心として質量分析による微生物同定技術の高度化に関する研究開発を推進するとともに、MALDI-TOF MSなどの質量分析機器の開発、製造、販売などを行っています。
今回、産総研が有する原核微生物大規模ゲノム情報解析技術と培養技術、島津製作所が有する質量分析技術と分析機器開発力を融合し、MALDI-TOF MSに基づく迅速微生物同定法のための解析プラットフォームの開発に至りました。
なお、本研究開発の一部は、国立研究開発法人日本医療研究開発機構(AMED)の委託事業「次世代治療・診断実現のための創薬基盤技術開発(腸内マイクロバイオーム制御による次世代創薬技術の開発)(2021~2026年度)(課題番号:22ae0121035h0002)」による支援を受けています。
今回の技術は、MALDI-TOF MSによる多様な微生物種同定のため、微生物ゲノム情報を利用した新しいアプローチを考案、原核微生物の大規模理論タンパク質量データベースの構築と、それを利用した解析アルゴリズムの開発で実現しました。本開発技術のコアとなる大規模理論タンパク質量データベースの構築にあたっては、公共の塩基配列データベースから原核微生物ゲノム情報を入手して、合計 約20万件のバクテリア・アーキアのゲノム情報を利用しました。これらは、およそ3.1万の種レベルの系統分類群を代表しています。本ゲノム情報をもとに、ゲノム中の遺伝子にコードされたタンパク質量を予測する独自の情報解析方法を構築し、個々の微生物ゲノム配列から全てのタンパク質の分子量を理論的に予測しました。それら理論分子量のリストが、従来法で培養微生物細胞を利用しMALDI-TOF MSで測定した質量スペクトル(ピークリスト)に相当します(図1)。本技術のゲノム情報から予測した理論タンパク質量データベースをGPMsDB(Genomically predicted protein mass database)と称しました。本データベースは、実測質量スペクトルから構築される質量データベースと同様に利用でき、かつそれらを置き換える可能性を持っています。
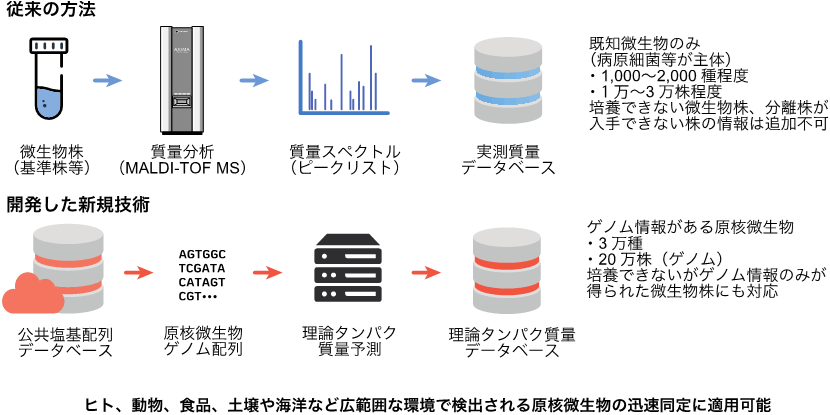
図1 従来方法との比較
ゲノム情報から予測した理論タンパク質量データベース(GPMsDB)により、従来不可能であった広範囲な原核微生物を質量分析により迅速同定することを可能にしました
同時に、膨大な理論タンパク質量情報をもとに正確、迅速な同定を行うための新規解析アルゴリズムを開発し、ソフトウェアとして実装しました(https://github.com/ysekig/GPMsDB-tk)。また、ユーザーが独自に入手した微生物ゲノム情報を入力として、GPMsDBに新しい微生物理論タンパク質量情報を追加する機能も付与しました(https://github.com/ysekig/GPMsDB-dbtk)。GPMsDB、関連するソフトウェアは公開しています。
本技術の分析妥当性の検証のため、15の異なる門レベル系統分類群を代表する94株(バクテリア84株、アーキア10株)の培養株による同定精度の評価を行いました。また、本研究以外で取得されたMALDI-TOF MSデータセット(独立行政法人製品評価技術基盤機構からの公表情報など)による同定精度の評価も併せて行いました。その結果、属レベル、種レベルでは90%以上の高い正答率が得られました。また、株レベルや亜種レベルでも高い正答率(70%以上)が得られており、種、あるいは亜種レベルでの微生物同定が可能な技術であることが明らかになりました。
さらに、メタゲノム解析と開発した微生物同定技術を融合した新たな網羅的培養(カルチャロミクス)のアプローチを考案(図2)、その検証を行いました。本アプローチは、培養の対象となるマイクロバイオーム検体をショットガンメタゲノム解析に供し、マイクロバイオームを構成する個々の微生物ゲノム配列を計算機上で再構築します。それらメタゲノム情報から得られたゲノム(metagenome-assembled genomes, MAGs)をGPMsDBに追加することで、生態系に存在する微生物株をMALDI-TOF MSで検出、同定できるようGPMsDBを拡張します。本アプローチは、同一試料より微生物の培養、分離を実施、MALDI-TOF MSと拡張したGPMsDBを利用した微生物を同定します。本研究では、ヒト腸内マイクロバイオームのカルチャロミクスによる有用微生物のスクリーニングを模擬し、マウスの糞便試料を利用しました。本試料に対しショットガンメタゲノム解析を実施し、構成ゲノムを再構築したところ、高い再現精度を持つ84種のゲノム配列情報が得られました。同一サンプルから嫌気性微生物の培養を実施して、100個以上のコロニーを選択することで、合計103個の分離培養液が得られました。それらに対してMALDI-TOF MSとGPMsDBを利用した同定を実施しました。その結果、属レベルでは103個のコロニー中94個が正確に同定されました。そのうち、12個の分離株は、本研究でのメタゲノム解析により得られたMAGsと同じ株であると同定されました。また、属、あるいは種レベルの未培養微生物が検出され、新しい微生物種が得られていることが明らかになりました。これらの結果は、カルチャロミクスにおいて、対象生態系のメタゲノム情報、例えばMAGなどゲノム情報から予測された機能遺伝子や代謝能などに基づく培養分離株のスクリーニングや、対象生態系には存在するが、まだ公共の塩基配列データベースに登録されていない新規未培養微生物群のスクリーニングなどの微生物探索に本技術が活用できることを示しています。
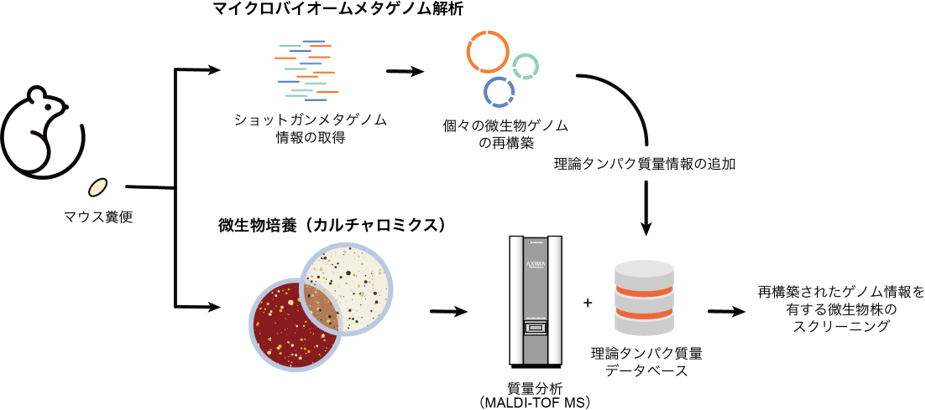
図2 メタゲノム解析と大規模理論タンパク質量情報を組み合わせた新しい微生物培養(カルチャロミクス)のスクリーニング、同定アプローチ
メタゲノム情報から得られたMAGsを理論タンパク質量データベース(GPMsDB)に追加することによりデータベースを拡張、同じ生態系のカルチャロミクスでのスクリーニング、同定精度を向上させます
本研究では、測定された質量スペクトルの90%以上について正しい微生物同定が達成されました。本研究で開発した技術群は、臨床検査や食品の安全性評価、環境診断などで実施される微生物検査に広く活用できる技術です。また、ヒトマイクロバイオームの研究開発分野で実施される、カルチャロミクスでの何千もの分離株の迅速なスクリーニングを可能にします。
今後は、より正確な同定を達成するためのアルゴリズムを開発するとともに、真菌などの異なる微生物群の同定が可能となるよう研究開発を実施します。また、公共塩基配列データベースに日々追加される微生物ゲノム情報を基により広範囲な原核微生物種の検出が可能となるようデータベースの充実を進めます。また、本開発技術をヒトマイクロバイオーム、とりわけ腸内マイクロバイオーム中の有用微生物群の網羅的な培養に活用し、ヒトの健康などに有用な微生物群の取得と産業利用を目指します。
掲載誌:Genome Biology
論文タイトル:A large-scale genomically predicted protein mass database enables rapid and broad-spectrum identification of bacterial and archaeal isolates by mass spectrometry
著者:Yuji Sekiguchi, Kanae Teramoto, Dieter M. Tourlousse, Akiko Ohashi, Mayu Hamajima, Daisuke Miura, Yoshihiro Yamada, Shinichi Iwamoto, Koichi Tanaka
DOI:10.1186/s13059-023-03096-4