横浜市立大学木原生物学研究所 清水健太郎 客員教授(チューリッヒ大学 教授兼任)および農業・食品産業技術総合研究機構(以下、農研機構) 半田裕一 ユニット長(現 京都府立大学 教授)、京都大学大学院農学研究科 那須田周平 教授、株式会社ヒューマノーム研究所 瀬々潤 代表取締役社長(産業技術総合研究所 招聘研究員兼任)らの研究グループが参加した世界10ヵ国から成る国際共同研究コンソーシアム「国際コムギ 10+ゲノムプロジェクト*1」は、世界各地で栽培されているコムギ15品種のゲノム解読に成功しました(図1)。日本チームは、日本を代表する実用品種「小麦農林61号」の解読に加え、進化ゲノム解析*2や染色体観察を担当しました。
パンコムギ(学名:Triticum aestivum L.)は、イネ・トウモロコシと並ぶ世界三大穀物ですが、実用品種のゲノム配列情報が不足しており、ゲノム配列の比較解析や、ゲノム情報を利用した現代的な分子育種への展開が遅れていました。今回、de novoゲノムアセンブリ*3というゲノム解析技術を用いることで、初めて、15品種について高精度のゲノム配列を得ることに成功しました。これにより分子育種技術の開発に欠かせない品種間差についての比較ゲノム・進化ゲノム解析が可能となりました。今後、ゲノム情報を活用したパンコムギの育種研究や品種改良が、国内外で飛躍的に進むと期待されます。その中でも農林61号は今回同時に解読された欧米の品種群とは大きく異なる遺伝的背景を持つため、アジアのコムギ品種の参照ゲノムとして広く利用されていくと考えられます。
*国際コムギ 10+ゲノムプロジェクトは、カナダ・サスカチュワン大学のカーティス・ポズニアック教授の統括の下で実施され、日本の研究チームとして、横浜市立大学木原生物学研究所、農研機構、京都大学の他、京都府立大学、産業技術総合研究所および(株)ヒューマノーム研究所の研究グループが参画しています。
世界三大穀物の一つであるパンコムギは、世界の人類が日常消費するカロリーの約2割を担っているとともに、タンパク質源としても人類の食料基盤を支えています。日本人の日常的な食生活にとっても、パン・うどん・菓子などに欠かせない極めて重要な作物です。しかし、近年の地球規模での気候変動により世界のコムギ生産は大きな影響を受けており、実際、2006年のオーストラリア大干ばつの際には日本でもコムギなどの価格が高騰し一般生活にも影響が及びました。また、将来日本でも暖冬によるコムギ減収がさらに進むと予想されており、温暖化などの気候変動をはじめさまざまな食料危機に対応できる新品種開発が急務と考えられています。
一般的にゲノム情報を利用した育種研究では、複数品種間でゲノムを比較して各品種の「個性」をもたらす遺伝子の違いをリストアップする必要があります。しかし、パンコムギについては、ゲノムサイズが16Gb(160億塩基)とヒトゲノムの4倍もあること、さらに3つの異なる種の交雑により倍数化した「異質六倍体*4」であることから、ゲノム構造が非常に複雑で解読が困難でした。これまでコムギでは、2018年に日本を含めた国際コンソーシアムによって実験系統Chinese Springのゲノム解読が行われたのみで(Science, 2018)、近代的な実用品種の配列の変異情報が欠けており、比較解析を十分に行うことができませんでした。そこで、複数の実用品種の高精度ゲノム配列を解読することで比較解析を可能にするプラットフォームの構築を目指してコンソーシアムが設立され、国際コムギ10+ゲノムプロジェクトが立ち上がりました。本プロジェクトの解析対象はほとんどが欧米の品種ですが、東アジア、特にコムギ伝播の東端である日本で独自に育種されてきたコムギは、その生育環境の大きな違いから遺伝的に他のコムギ品種と大きく異なることが予想され、有用な資源として注目されていたことから、コンソーシアムからの強い要望により、今回の日本の研究チームの参加と、日本品種のゲノム解読を行うことが決まりました(図1)。
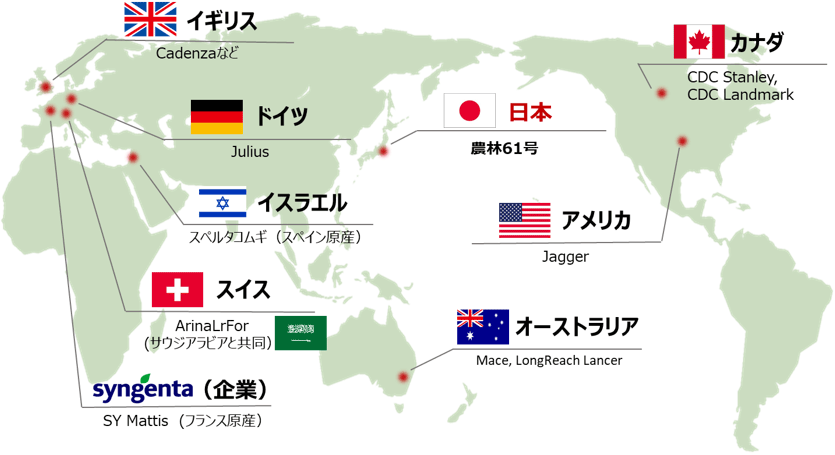
図1:国際コムギ10+ゲノムプロジェクトで解読した主なコムギ品種と参加国
国際コンソーシアムにより、世界の代表的なコムギ15品種の高精度ゲノム解読を行った。
日本の研究チームは、「農林61号」の解読を担当した。
本プロジェクトでは、世界各国の代表的なコムギ品種の中からパンコムギ近代品種14品種と、古くから栽培されてきたスペルタコムギ(学名:Triticum spelta L.)1品種の計15品種が選ばれました。日本の研究チームは、20世紀半ばから半世紀にわたり日本で広く栽培され、京都大学が運営しているナショナルバイオリソースプロジェクトで保存されていた日本の代表的パンコムギ品種「農林61号」のゲノム解読を行いました(図1、2)。
これまでのゲノム研究では、一系統だけを高精度で解読した配列を標準とし、その他の系統は低い精度で解読して比較することが一般的でした。しかしながら、この手法では初めに解読した系統に存在しない遺伝子の解析ができないという欠点があります。今回、国際共同研究でデータ・解析手法を集積することにより、標準となるゲノムの情報を使わないde novoゲノムアセンブリという手法が可能になり、この新手法を用いて農林61号をはじめそれぞれの品種のゲノムの解読を行いました。その結果、これまでに唯一ゲノム解読が完了していた実験系統Chinese Springでは、例えば病原抵抗性に関わるNLR遺伝子群は約 2,500遺伝子見つかっていましたが、さらに今回のプロジェクトで2,500以上の新しい遺伝子が見つかりました。

図2:日本チームが解読を担当した農林61号
九州から北海道まで広い地域で半世紀以上にわたって栽培されてきた日本を代表する品種。
本プロジェクトでは、京都大学が保存してきた農林61号の系統種子を使用した。現在、ゲノム情報を解読した系統として新しい系統番号LPGKU2305を与えられてナショナルバイオリソースプロジェクト(NBRP)・コムギに保存され、各国で栽培試験が行われている。
(左)農林61号の穂(提供:木原生物学研究所)
(右)カナダで試験栽培される農林61号
さらに日本の研究チームは、得られた15品種のゲノム配列データを基に独自の解析を行い、次のような新たな知見を得ることに成功しました。
- 極東にある日本品種の農林61号は、今回解読された欧米の他の品種と比べてゲノムDNA配列が大きく異なることを明らかにしました(図3)。
- 異質六倍体であるパンコムギはそれぞれの遺伝子が3コピーずつあります。DNA変異のパターンを調べたところ、増加した遺伝子コピーがそれぞれ異なった進化の歴史をたどってきたことがわかり、こうした遺伝子コピー間での機能分担が環境頑健性に貢献した可能性が示されました。
- 高精度ゲノム解読を、染色体DNA観察技術と組み合わせることにより、近代育種の過程で異なった染色体構造変異を持つ品種が選抜されてきたことが明らかになりました(図4)。他にも、育種の歴史の中で、野生種の染色体の大きなブロックがパンコムギに取り入れられてきたこともわかりました。ゲノム比較解析によって、農耕の歴史とともに染色体に刻まれたコムギの進化の歴史が見えてきました。
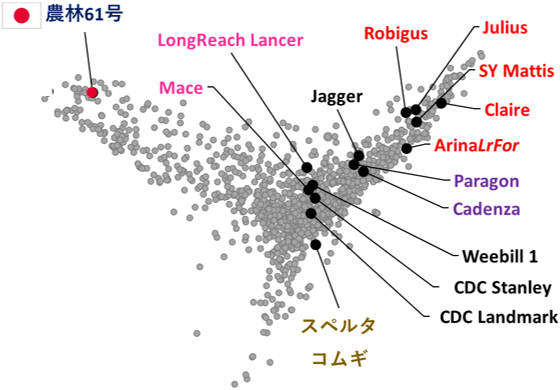
図3:品種間の遺伝的距離を2次元上に示した主成分分析結果
農林61号(赤丸)は、今回解析された14品種(黒丸)と大きく離れており、遺伝的に大きく異なることが見てとれる。今回解析された15品種と、過去にゲノムの一部の解析が行われた約1,200品種(灰色)の遺伝的多型情報を合わせて解析を行った。
品種名の色は原産地などの特徴を示している。
青:アジア
桃:オーストラリア
黒:南北アメリカ
赤:ヨーロッパの秋播性コムギ
紫:ヨーロッパの春播性コムギ
黄:ヨーロッパの在来品種
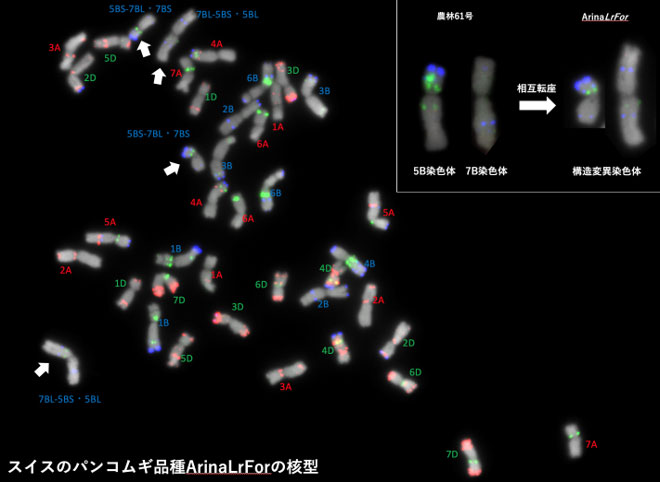
図4:パンコムギ品種に見つかった大規模構造変異の一例
3種類の局在型反復配列(図中ではそれぞれ、赤、青、緑に擬似的に着色されている)の局在パターンの比較によって、染色体構造変異を分子細胞遺伝学的に確認した。スイスの品種ArinaLrForは5B染色体と7B染色体間の転座染色体を持つ(図中で白矢印)。5B染色体短腕と7B染色体長碗間の相互転座がゲノム配列比較でも核型分析でも支持された(右上挿図)。

図5:パンコムギの祖先種を発見した木原均博士
(提供:木原生物学研究所)
今回の成果により、農林61号をはじめさまざまなコムギ品種の遺伝情報を利用した、より迅速な品種開発やテーラーメイド育種が可能となります。国際コムギ10+プロジェクトは、今後さらに100ゲノム、1,000ゲノムへの展開が計画されており、今回の研究で得られた精度の高いコムギのゲノム配列は、今後、あらゆる基礎研究・育種応用研究共通の「基準」として、幅広くかつ将来にわたって長く活用されることは間違いありません。
これまで日本は、遺伝学者・木原均(図5)の伝統を受け継ぎ、世界のコムギ研究をリードしてきました。日本などアジアのコムギ遺伝資源は欧米品種とは異なる特性を持っており、20世紀半ばの「緑の革命*5」などで世界の穀物増産に貢献してきました。一方で、いまだ世界のコムギ育種に十分活用されていないことも指摘されています。今回、農林61号は欧米の他品種と比較してゲノムDNA配列の違いが大きく特徴的であることが全ゲノムレベルで浮き彫りになりました。この農林61号は、日本の九州から北海道まで広い地域で栽培可能な広域適応性、つまり環境頑健性を持っています。今回の農林61号のゲノム解読によって、アジアのコムギの持つ優れた形質が、今後、日本だけでなく世界中の国々でコムギの環境頑健性を高める研究と育種に活用され、第2の「緑の革命」を起こし得る優良品種の作出、さらには持続可能な開発目標(SDGs)達成に向けた食料安全保障に大きな役割を果たすものと期待されます。
Multiple wheat genomes reveal global variation in modern breeding
Sean Walkowiak, Liangliang Gao, Cecile Monat, Georg Haberer, Mulualem T. Kassa, Jemima Brinton, Ricardo H. Ramirez-Gonzalez, Markus C. Kolodziej, Emily Delorean, Dinushika Thambugala, Valentyna Klymiuk, Brook Byrns, Heidrun Gundlach, Venkat Bandi, Jorge Nunez Siri, Kirby Nilsen, Catharine Aquino, Axel Himmelbach, Dario Copetti, Tomohiro Ban, Luca Venturini, Michael Bevan, Bernardo Clavijo, Dal-Hoe Koo, Jennifer Ens, Krystalee Wiebe, Amidou N’Daiye, Allen K. Fritz, Carl Gutwin, Anne Fiebig, Christine Fosker, Bin Xiao Fu, Gonzalo Garcia Accinelli, Keith A Gardner, Nick Fradgley, Juan Gutierrez-Gonzalez, Gwyneth Halstead-Nussloch, Masaomi Hatakeyama, Chu Shin Koh, Jasline Deek, Alejandro C. Costamagna, Pierre Fobert, Darren Heavens, Hiroyuki Kanamori, Kanako Kawaura, Fuminori Kobayashi, Ksenia Krasileva, Tony Kuo, Neil McKenzie, Kazuki Murata, Yusuke Nabeka, Timothy Paape, Sudharsan Padmarasu, Lawrence Percival-Alwyn, Sateesh Kagale, Uwe Scholz, Jun Sese, Philomin Juliana, Ravi Singh, Rie Shimizu-Inatsugi, David Swarbreck, James Cockram, Hikmet Budak, Toshiaki Tameshige, Tsuyoshi Tanaka, Hiroyuki Tsuji, Jonathan Wright, Jianzhong Wu, Burkhard Steuernagel, Ian Small, Sylvie Cloutier, Gabriel Keeble-Gagnere, Gary Muehlbauer, Josquin Tibbets, Shuhei Nasuda, Joanna Melonek, Pierre J. Hucl, Andrew Sharpe, Matthew Clark, Erik Legg, Arvind Bharti, Peter Langridge, Anthony Hall, Cristobal Uauy, Martin Mascher, Simon G. Krattinger, Hirokazu Handa, Kentaro K. Shimizu, Assaf Distelfeld, Ken Chalmers, Beat Keller, Klaus F. X. Mayer, Jesse Poland, Nils Stein, Curt A. McCartney, Manuel Spannagl, Thomas Wicker, and Curtis J. Pozniak
Nature(2020). https://doi.org/10.1038/s41586-020-2961-x
※同時発表(11月27日オンライン予定)
De Novo Genome Assembly of the Japanese Wheat Cultivar Norin 61 Highlights Functional Variation in Flowering Time and Fusarium Resistance Genes in East Asian Genotypes
Kentaro K. Shimizu, Dario Copetti, Moeko Okada, Thomas Wicker, Toshiaki Tameshige, Masaomi Hatakeyama, Rie Shimizu-Inatsugi, Catharine Aquino, Kazusa Nishimura, Fuminori Kobayashi, Kazuki Murata, Tony Kuo, Emily Delorean, Jesse Poland, Georg Haberer, Manuel Spannagl, Klaus F. X. Mayer, Juan Gutierrez-Gonzalez, Gary J. Muehlbauer, Cecile Monat, Axel Himmelbach, Sudharsan Padmarasu, Martin Mascher, Sean Walkowiak, Tetsuya Nakazaki, Tomohiro Ban, Kanako Kawaura1 Hiroyuki Tsuji, Curtis Pozniak, Nils Stein, Jun Sese, Shuhei Nasuda, Hirokazu Handa
Plant and Cell Physiology(2020). https://doi.org/10.1093/pcp/pcaa152
※本研究は、科学技術振興機構(JST)戦略的創造研究推進事業 CREST「環境変動に対する植物の頑健性の解明と応用に向けた基盤技術の創出」、文部科学省科研費 新学術領域研究「植物新種誕生の原理」、農研機構、産業技術総合研究所、日本医療研究開発機構(AMED)「ナショナルバイオリソースプロジェクト(NBRP)」などの支援を受けて遂行されました。