独立行政法人 産業技術総合研究所【理事長 吉川 弘之】(以下「産総研」という)生物機能工学研究部門【研究部門長 巌倉 正寛】機能性核酸研究グループ 富田 耕造 研究グループ長、渡邉 和則 産総研特別研究員、董 雪松 産総研特別研究員、須藤 恭子 研究員らは、国立大学法人 東京大学の協力を得て、遺伝暗号なしにアミノ酸をタンパク質に連結する酵素(LF転移酵素)の反応機構を明らかにした。
これにより、新規タンパク質合成や機能性タンパク質開発技術、この酵素系をターゲットにした新たな抗細菌薬剤のデザイン等が期待される。
本研究成果は、平成19年9月23日18時(英国時間)に、英国科学誌 Nature 電子版に掲載される。
細胞内における遺伝情報の通常の流れはDNA→mRNA→タンパク質であり、タンパク質は20種類のアミノ酸が遺伝暗号にしたがって連結されて合成される。細胞内でのアミノ酸同士の連結反応は、リボゾームと呼ばれるタンパク質合成装置内で行われる。リボゾームではmRNAの遺伝暗号にしたがって必要なアミノ酸がtRNAによってつぎつぎと運ばれてきて結合される。
これとは別にリボゾーム外で、tRNAによって運ばれて来たアミノ酸を直接細菌の細胞壁の構成成分や、細胞内のタンパク質に付加結合させる酵素群が40年以上前から知られていた。また、リボゾーム外でアミノ酸をタンパク質へ付加結合する同様な酵素は、ヒトをはじめ、ほとんどの生物にも見出されており、応用面からも重要である。しかしながら、その付加結合反応の機構については長年の謎であった。
リボゾーム外での反応機構の解明は、タンパク質合成系の進化、構築原理を探る上で興味深く、産総研では、核酸を用いないRNA合成酵素の研究、遺伝暗号を介さないタンパク質合成酵素の研究等、細胞内における通常の遺伝情報の流れ(DNA→mRNA→タンパク質)から外れた現象に注目して研究してきた。
本研究では、遺伝暗号に依存しないでアミノ酸の結合反応を触媒する酵素の反応機構を明らかにするため、酵素とタンパク質、あるいはアミノ酸、tRNA、酵素の複合体のX線結晶構造解析、さらにその構造を基にした生化学的解析を行った。
本研究は、独立行政法人 日本学術振興会、文部科学省および財団法人 倉田記念日立科学技術財団からの研究補助金によって行われた。国立大学法人 東京大学には構造解析に用いた試料の有機合成について協力を得た。また、X線結晶回折実験は、大学共同利用機関法人 高エネルギー加速器研究機構 物質構造科学研究所の放射光科学研究施設のビームラインBL-5A、AR-NW12、NW-17を使用した。
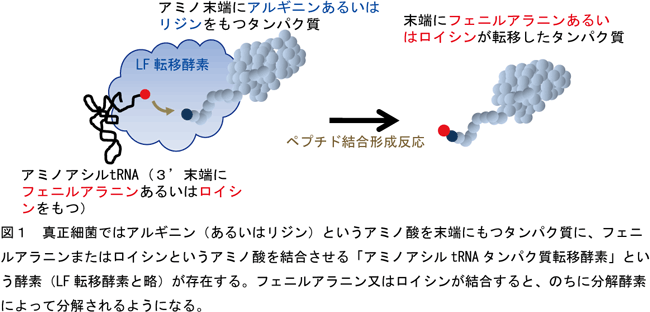
生体内で合成されたタンパク質はいつまでも存在し続けるのではなく、不要になった時には分解されなければならず、分解のされやすさは厳密にコントロールされている。大腸菌のような真正細菌ではアルギニン(あるいはリジン)というアミノ酸を末端にもつタンパク質に、フェニルアラニン又はロイシンというアミノ酸が結合すると、のちに分解酵素によって分解されるようになる。このフェニルアラニン又はロイシンというアミノ酸をタンパク質末端に結合させる反応はリボゾームを介さないで行われ、tRNAによって運ばれてきたフェニルアラニン又はロイシンを直接タンパク質の末端に転移して結合を形成する。この結合反応は「アミノアシルtRNAタンパク質転移酵素」という名前の酵素(以下LF転移酵素と略)によってなされる(図1)。
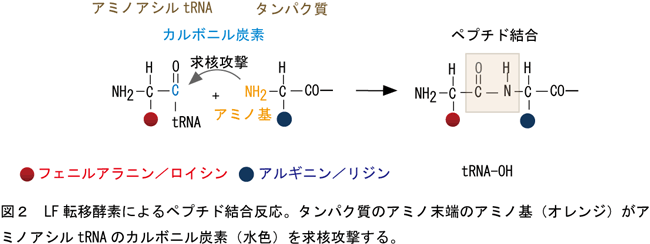
この酵素はタンパク質の基本骨格であるペプチド結合をリボゾーム外で形成するという点で非常にユニークである。結合反応はタンパク質のアミノ末端のα-アミノ基(NH2)がアミノアシルtRNAのカルボニル炭素(C)を攻撃する反応であるが(図2)、これら2つの物質を混ぜただけでは反応は進まず、酵素がどのようにこの反応を進めるのかわかっていなかった。
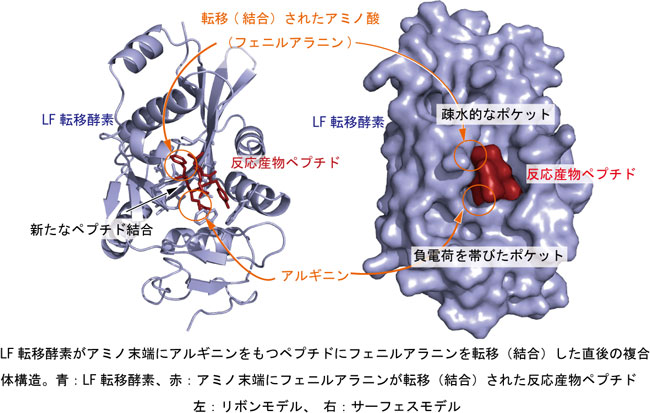
今回、LF転移酵素だけでなく関係する複合体(下記(1)と(2))のX線結晶構造解析を行った。
(1)tRNAにフェニルアラニンが付加したアミノアシルtRNAの末端部分のモデルであるフェニルアラニルアデノシン(rA-Pheと以下略)と酵素の複合体。
(2)タンパク質の末端アミノ酸であるアルギニンのアミノ末端にフェニルアラニンが転移結合された短いモデルペプチド(アミノ酸配列はフェニルアラニン-アルギニン-チロシン-ロイシン-グリシン)と酵素の複合体。この短いペプチドと酵素の複合体構造はペプチド結合反応が終了した直後の構造を表している。
これらの2つのX線結晶構造解析による構造の重ね合わせから、以下のことが明らかになった(図3)。
|
1) |
rA-Pheのフェニルアラニンは酵素の疎水的なポケットによって認識されている。(フェニルアラニン、ロイシン、イソロイシンのような疎水的なアミノ酸に囲まれている。) |
|
2) |
ペプチドのアルギニンは疎水的なポケットに隣接する負電荷(電気的に陰性)を帯びたポケットによって認識されている。(グルタミン酸のような負電荷アミノ酸と相互作用している。) |
|
3) |
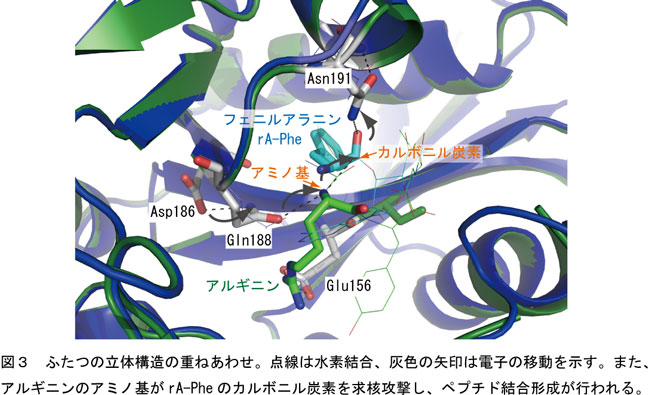
ペプチドのアルギニンのα-アミノ基がrA-Pheのカルボニル炭素と近いところに位置し、カルボニル炭素を求核置換攻撃ができる場所に位置している。 |
|
4) |
アルギニンのα-アミノ基はGln188と近いところに位置し水素結合を形成している。 |
|
5) |
Gln188はAsp186と水素結合をし、Asn191はrA-Pheのカルボルニ基と水素結合をしている。 |
(注:Asp186はタンパク質N末端から186番目のアミノ酸アスパラギン酸、 Gln188は188番目のアミノ酸グルタミン、Asn191は191番目のアミノ酸アスパラギンのこと)
また、酵素の活性触媒に重要と考えられるAsp186、Gln188、さらにAsn191、またアルギニンの側鎖を認識するGlu156をアラニンへ置換した変異体酵素を作製し、それら酵素の反応速度の解析も行った。その結果、いずれの変異体酵素でもその活性がほとんど失われた。(注:Glu156はタンパク質N末端から156番目のアミノ酸グルタミン酸のこと。)
X線結晶構造解析、および構造を基にした活性に関与するアミノ酸を置換した上記変異体酵素の速度論解析から、この酵素の反応機構について次のようなステップで進むことがわかった。
|
1) |
Asp186からGln188への電子移動によって、Gln188が電気的に陰性になりアルギニンのα-アミノ基から電気的に陽性の水素原子が引きつけられる。 |
|
2) |
電気的に陽性の水素が遠ざかることによってα-アミノ基の窒素(N)原子は電気的に陰性が強く電子リッチになる。 |
|
3) |
電子リッチに(陰性)なった窒素(N)原子は、アミノアシルtRNAの電子プアーな(電気的に陽性)カルボニル炭素(C)を求核攻撃する。 |
|
4) |
この攻撃によってフェニルアラニンのカルボニル炭素(C)とアルギニンのα-アミノ基の窒素(N)が結合してペプチド結合ができる。また、求核置換反応の際に生じる正四面体中間状態はAsn191によって安定化されていることが明らかになった(図4)。 |
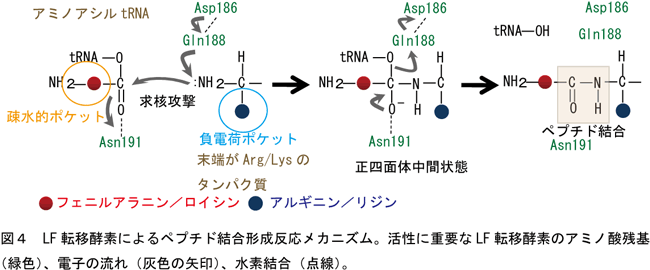
この研究で明らかになったLF転移酵素によるペプチド結合形成の反応触媒機構は、多種類のRNAやタンパク質が関与するリボゾームでのペプチド結合形成反応機構とは全く異なる新規の反応である。
今後、この酵素の改変体を用いたリボゾームを介さない新規タンパク質合成システムの開発や、非天然アミノ酸をタンパク質へ導入し、機能性タンパク質を合成するシステムの開発を行う予定である。また、細菌由来のこれらの酵素の新たな反応機構に基づいた特異的な阻害剤の開発も期待される。