独立行政法人 産業技術総合研究所【理事長 吉川 弘之】(以下「産総研」という)生物機能工学研究部門【部門長 巌倉 正寛】のP.K.R. Kumar(ペンメチャ K.R. クマール)主任研究員らは、独立行政法人 農業生物資源研究所【理事長 岩渕 雅樹】(以下「生物研」という)と、枯草菌のL-ヒスチジン分解酵素遺伝子であるhut構造遺伝子の発現機構は、転写制御蛋白質HutPが標的のRNAに結合することで、RNAがステム-ループ構造から新規の三角形に構造変化し、遺伝子のスイッチをONにする機構であることを解明した。本研究はhut構造遺伝子発現の際に形成されるアンチターミネーション複合体(転写制御蛋白質がRNAに結合することにより起こる遺伝子転写制御の一つで、転写抑制を解除するときに生じる複合体)を結晶化し、そのX線結晶構造解析により転写制御蛋白質HutP、RNAの構造変化の過程を明らかにしたもので、これにより、遺伝子の制御機構の研究が加速されるものと期待される。
これまで、転写制御蛋白質とRNAとの複合体の構造研究では、転写制御蛋白質とその転写制御蛋白質を活性化させる分子であるリガンドの結合前と結合後の転写制御蛋白質の構造変化が調べられていなかった。本研究では、転写制御蛋白質とリガンドであるL-ヒスチジンとMg2+イオンが結合する前と結合後の転写制御蛋白質及びRNAの構造変化をX線結晶構造解析により分子レベルで解明を行った。
枯草菌は、飢餓状態になるとL-ヒスチジン分解酵素遺伝子であるhut構造遺伝子が発現してL-ヒスチジンを分解し、生成した炭素と窒素をエネルギー源として使用する。このhut構造遺伝子は、5個の構造遺伝子hutH, hutU, hutI, hutG, hutMから構成され、hut構造遺伝子の発現は、転写制御遺伝子hutPによって制御されている。hut構造遺伝子と転写制御遺伝子hutPは、一つながりの遺伝子(hut遺伝子)であり、一つの転写単位であることからhutオペロンと呼ばれている。転写制御遺伝子hutPとhut構造遺伝子の間に位置するRNA配列はステム-ループを形成し、ターミネーター機能をもつ。すなわち、ステム-ループの下流にあるhut構造遺伝子の発現を抑制している(通常状態)。しかし、飢餓状態では、転写制御遺伝子hutP の産物である転写制御蛋白質HutPは、L-ヒスチジン、Mg2+イオンにより活性化されることにより、ステム-ループと特異的に結合し、ステム-ループの構造変化を引き起こし、アンチターミネーション複合体を形成する。これによりステム-ループがもつターミネーター機能は消失する。
これまでの研究から、このRNA、つまりステム-ループの構造変化が、下流のhut構造遺伝子の転写のスイッチをONにするものと推測されており、本研究では、転写制御蛋白質HutP単独からアンチターミネーション複合体に至るまでの中間体構造のX線結晶構造解析を行い、結合したRNAはステム-ループ構造から新規の3角形に構造変化したことを実証した【図1参照】。この結果から、hut構造遺伝子の発現機構は、転写制御蛋白質HutPの介在により引き起こされたRNAの構造変化が、下流のhut構造遺伝子の発現のスイッチをONにする機構であることが明らかになった。
今回のアンチターミネーション複合体のX線結晶構造解析、及び中間構造のX線結晶構造解析によって、転写制御蛋白質HutP及び標的のRNAの構造変化をとらえることに成功した。今後は、遺伝子及びそれを制御する蛋白質の構造変化と遺伝子のスイッチをON、OFFにする制御機構について、枯草菌以外の他の原核生物にも範囲を広げ、引き続き研究を進めていく予定である。
本研究成果は、英国科学誌 Nature 2005年3月10日号* に掲載された。
*Kumarevel, T., Mizuno, H. and Kumar, P.K.R. (2005) Structural basis of HutP-mediated anti-termination and roles of the Mg2+ ion and L-histidine ligand. Nature 434, 183-191 (2005)
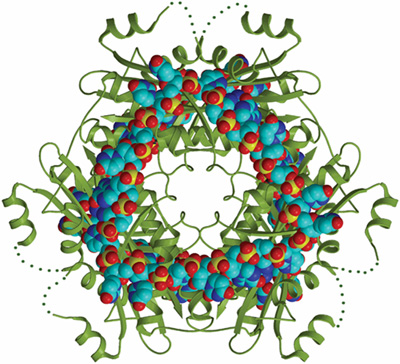 |
図1.アンチターミネーション複合体の立体構造
緑色のリボンモデルが転写制御蛋白質HutPで、赤、水色、青等のボール状で連なった3角形の形をしたものがRNA(図3[4]の三角形に該当)である。 |
遺伝子発現は転写、翻訳の過程を経て行われる。それらの過程はDNA又はRNAと様々な蛋白質の相互作用によりコントロールされている。これまで転写制御に関与するアンチターミネーションの構造生物学的研究の報告はあるが、これら転写制御蛋白質とRNAのアンチターミネーション複合体では、転写制御蛋白質とその転写制御蛋白質を活性化させる分子であるリガンドの結合前と結合後の転写制御蛋白質の構造変化が調べられていない。これまでの研究で、リガンドにより構造変化した転写制御蛋白質がRNAに結合し、その結合によって引き起こされたRNAの構造変化がmRNAの転写のスイッチをONにすると推測されており、本研究ではX線結晶構造解析により、その分子レベルでの解明を行った。
産総研 生物機能工学研究部門のP.K.R. Kumar主任研究員らは、以前より新しい機能をもつRNA分子の創製や、遺伝子発現の過程で生じるRNAと蛋白質との特異的相互作用に関連した研究を展開しており、多くの国際的な成果をあげている。RNAと蛋白質の相互作用研究には分子構造の解明が必須であることから、X線結晶構造解析の分野で成果をあげている生物研 生体高分子研究グループの 水野 洋 研究チーム長(現;NECソフト(株)主席研究員)と研究を行ってきた。
なお、本研究成果において、生物研では、文部科学省の科学技術振興調整費「開放的融合研究プロジェクト」の支援を受けている。
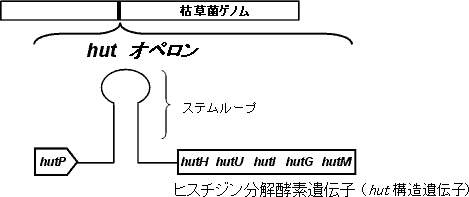
枯草菌は、飢餓状態になるとL-ヒスチジン分解酵素遺伝子であるhut構造遺伝子が発現してL-ヒスチジンを分解し、生成した炭素と窒素をエネルギー源として使用する。このhut構造遺伝子は、5個の構造遺伝子hutH, hutU, hutI, hutG, hutMから構成され、hut構造遺伝子の発現は、転写制御遺伝子hutPの産物である転写制御蛋白質HutPによって制御されている。hut構造遺伝子と転写制御遺伝子hutPは、一つながりの遺伝子(hut遺伝子)であり、一つの転写単位であることからhutオペロンと呼ばれている【図2参照】。
転写制御遺伝子hutPとhut構造遺伝子の間に位置するRNA配列はステム-ループを形成し、通常はターミネーター(遺伝子の発現を抑制している)機能を発現している。飢餓状態ではこのターミネーター機能は、L-ヒスチジンと結合した転写制御蛋白質HutP(hutP産物)によって解除される。すなわち転写制御蛋白質HutPは、L-ヒスチジン、Mg2+イオンの存在下で上述のステム-ループ領域のRNAと特異的に結合し、そのRNAの構造変化を引き起すことによりアンチターミネーション複合体を形成している。これまでの研究から、このRNA、つまりステム-ループの構造変化が、下流のhut構造遺伝子の転写のスイッチをONにするものと推測されていたが、その分子レベルでの実証はなされていなかった。
本研究では、このアンチターミネーション複合体【図3[4]参照】のX線結晶構造解析、及びこの複合体からRNAを除いた複合体【図3[3]参照】、転写制御蛋白質HutPとL-ヒスチジンとの複合体【図3[2]参照】、複合体を形成しない転写制御蛋白質HutP単独のもの【図3[1]参照】のX線結晶構造解析を行うことにより、アンチターミネーションに至る中間構造を調べた。その結果、転写制御蛋白質HutPは6量体を形成していること【図3[1]参照】、L-ヒスチジンとMg2+イオンが加わると転写制御蛋白質HutPは構造変化すること【図3[2][3]参照】、構造変化した転写制御蛋白質HutPは標的RNA(RNAを構成する塩基U(ウラシル)A(アデニン)G(グアニン)の繰り返し配列をもつ)を認識し、特異的に結合すること【図3[4]参照】、結合したRNAは転写制御蛋白質HutP6量体の表面において新規の3角形の構造をとること【図3[4]参照】等が明らかになった。アンチターミネーションの形成過程で転写制御蛋白質HutP、RNAが巧妙に構造変化している様子が明らかになり、これらの結果から、hut構造遺伝子の発現機構は、転写制御蛋白質HutPの介在により引き起こされたRNAの構造変化が下流のhut構造遺伝子の発現のスイッチをONにする機構であることが明らかになった。
 |
|
図2.枯草菌hutオペロン
|
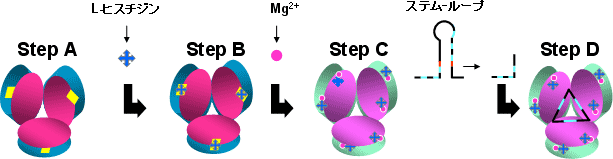 |
|
|
L-ヒスチジンが認識され、疎水性ポケットに取り込まれる |
さらに、Mg2+を取り込むとHutP6量体は構造変化する |
標的RNAと結合しアンチターミネーション複合体を形成。RNAは三角形に構造変化 |
|
[1]
|
[2]
|
[3]
|
[4]
|
図3.アンチターミネーション形成機構の模式図 |
今後は、遺伝子及びそれを制御する蛋白質の構造変化と遺伝子のスイッチをON、OFFにする制御機構について、枯草菌以外の他の原核生物にも範囲を広げ、引き続き研究を進めていく予定である。