独立行政法人 産業技術総合研究所【理事長 野間口 有】(以下「産総研」という)バイオメディシナル情報研究センター【研究センター長 嶋田 一夫】分子システム情報統合チーム 今西 規 招聘研究員らは、国立大学法人 総合研究大学院大学先導科学研究科 颯田 葉子 教授と共同で、ヒトおよび類人猿の進化の歴史を、全ゲノム配列情報を用いて解明した。
生物種のゲノム配列同士を比較し、配列間の違いの程度を集団遺伝学理論に基づき解析することによって、生物種がどのように進化してきたかを再構築することができる。本研究は、ヒト、チンパンジー、ゴリラ、オランウータンの全ゲノム配列を解析し、ヒトとチンパンジーの種が分岐したのは一度のみ起きたことを示し、「祖先におけるヒトとチンパンジーの交雑」仮説を否定した。また、ヒト系統とチンパンジー、ゴリラの種分岐年代はそれぞれ600~760万年前、760~970万年前と推定され、近年考えられていた分岐年代より古いことがわかった。これらの種分岐年代は、ヒトの進化研究においてゲノム配列および化石記録を統合して理解していく上で、決定的な指標になると期待される。この研究には膨大な計算量を要したため、2011年に産総研に導入された大型計算機AIST Super Cloudを用いた。
なお、この研究成果は、2012年9月12 日にGenome Biology and Evolutionにオンライン掲載され、同誌2012年11月号に掲載された。(Genome Biology and Evolution. 4:1133-1145, 2012)
 |
|
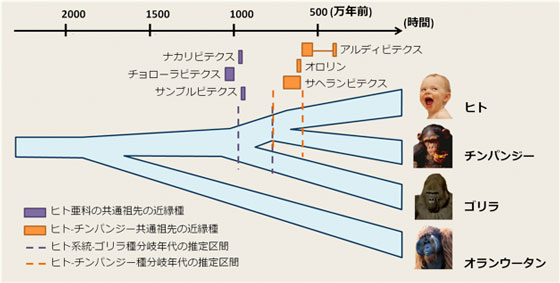
図1 ヒトと類人猿の進化の歴史 |
最初のヒトゲノム解読から11年経った2012年、ヒトゲノムが持つ機能を網羅的に調べ上げるプロジェクトENCODEの成果が発表された。今後は、ヒトゲノムを中心としたヒト疾患研究や創薬研究が加速されていくと期待される。
このようにヒトゲノムを産業的に活用することの基礎的な背景となるのが、「ヒトらしさ」の特徴の解明である。「ヒトらしさ」をかたちづくる遺伝的な特徴は、進化においてヒトの系統特異的に起きたゲノム上の変異により生じた。生活習慣病など多くの疾病に関する遺伝的変異がヒト系統に特異的に起きたことが知られているため、疾病がどのように生じたかを明らかにするにはヒト特異的なゲノムの変異とヒトに特有の特徴における変化の関係を明らかにする必要がある。
この「ヒトらしさ」をゲノムから理解するためには、ヒトの進化的な歴史をはじめに知らなければならない。遺伝的変異が起きた系統の特徴を明らかにすることにより、その変異がヒトにのみ起こったことかどうかが判断できるからである。しかしながら、ヒトと類人猿の種が分かれた過程については未だ統一見解が得られていない。古生物学および集団遺伝学では、ヒトと最も近縁種であるチンパンジーの間に種が分岐した後に交雑が起きたかについて長く論争になっており、2006年にはパターソン(Patterson)らが限られたゲノム配列を用いて交雑仮説を提唱している。一方で交雑を反証する論文も多く存在する。近年、ヒトゲノムだけではなく類人猿のゲノムも全長が解読され、網羅的なゲノムデータを用いてヒトとチンパンジーの種分岐問題を再検討できる条件が整って来ていた。
産総研では、ヒト完全長cDNAに基づくヒト遺伝子データベースH-InvDBを2004年に開発した。H-InvDBはヒト遺伝子研究において最初に参照すべきデータベースとして研究者間で長く支持されている。また、H-InvDBにはヒト遺伝子に関する様々な副次的なデータベースがあり、比較ゲノムデータベースG-compassや分子進化データベースEvolaでは、H-InvDBの成果をもとに解析、注釈づけされたゲノムおよび遺伝子の進化情報を公開している。本研究ではこれらのデータベースおよびデータ作成ツールを活用した。
なお、本研究はJSPS科研費(24657168)による支援を受けて行われた。
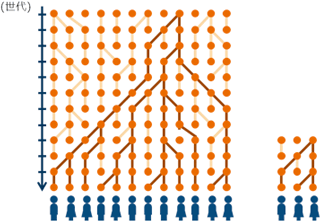
ゲノム配列は生物が持つ遺伝情報の全てであり、細胞内ではDNAとして存在し「生命の設計図」ともいわれる。広辞苑200冊分の情報(約30億文字)を持つヒトゲノム配列は、DNA複製においてごくまれに起きるエラーにより、親から子へ伝わる際に数十か所の「誤記」が生じる。このような誤記を突然変異と呼ぶ。長い年月の後に変異を持つ系統が途絶えてしまうこともあるが、ごくまれに集団が特定の変異を持つ子孫のみで構成されるようになることがある(固定という)(図2)。動物ゲノムにおける突然変異の大部分はゲノムが持つ生物としての機能には影響を与えないため、突然変異がその集団に固定するかどうかは偶然に決まることになる。一方、時々生じる有害な変異は、遺伝子の機能に支障をきたし子孫を残すことに不利になるため速やかに途絶えてしまい、さらにごくまれに生じる生存に有利な変異は集団に固定されやすい(これらを自然選択という)。変異と固定の過程を繰り返してゲノムそして生物は進化していく。
一方、共通祖先から進化してきたと考えられている現生生物種のゲノムは、共通祖先のゲノムに変異が蓄積された結果であると考えられる。従って、現生生物種間でゲノム配列を比較し、互いに異なるゲノム領域を判定することにより、それぞれの種が共通祖先から進化してきた歴史を推定できる。たとえば、ゲノム配列の差異が大きい種同士は古くに分かれ、差異が小さい種同士は近縁で、新しい時期に分かれたことがわかる。ゲノム配列の差異と分岐した時間の関係について統計モデルに当てはめることにより(図2)、種間の種分岐年代を推定することが可能になる。これまで長く用いられてきた骨や歯などの古代生物の化石記録から生物の進化を探る方法とは大きく異なり、ゲノム配列を用いて進化を推定する方法は、大量の情報と定量性に裏付けられる。
 |
|
図2 変異が集団に固定する過程
丸が集団中の個体、線が遺伝子の系統を意味する。濃色の系統が集団中に固定される。大部分を占める淡色の系統は途中で集団から消え集団には固定されない。この図を時間に対し逆にたどると、集団内の個体が持つ遺伝子は共通祖先を有していることがわかる。一方小さい集団(右図)では、変異が集団に広まる時間(あるいは現在から共通祖先までさかのぼる時間)は短くなる。 |
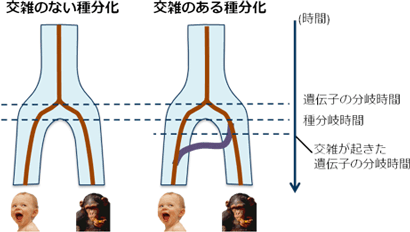
パターソン(Patterson)らは2006年にヒトとチンパンジーのゲノムの部分領域を用いた解析から、性染色体であるX染色体における種分岐年代は常染色体より新しいと推定し、その理由をヒトとチンパンジーが祖先において交雑したからであると発表した(図3)。交雑によりチンパンジーゲノムがヒトゲノムに混じると、そのゲノム領域の分岐年代は、最初に種が分岐した時期ではなく、交雑が起きた時期として推定される。このような交雑の痕跡はX染色体だけでなく全ての染色体に見られるはずであるが、パターソンらの研究は、交雑の痕跡がX染色体の全域に見られる一方常染色体には存在しないことを示している。パターソンらは、交雑が起きたヒトの祖先では、チンパンジーの祖先から受け継いだX染色体が生存に大きく有利にはたらき、チンパンジーの祖先のX染色体全域を持つ個体のみが生き残った(選択された)と説明した。
 |
|
図3 交雑の有無に基づく種分岐のモデル
交雑が無い場合には一回だけ種分岐が起きた(左図)。種分岐直前の集団にも図2のような系統関係が成り立っており、すなわち遺伝子(茶線)は種分岐に先駆けて(あるいは同時に)分岐したことを意味する。交雑がある場合(右図)には、種分岐の後に遺伝子の混入が起き(紫線)種分岐より新しい遺伝子の分岐が一部に観察される。 |
パターソンらの仮説には2つの疑問が残った。1つは、上記のように交雑の説明があまりにも複雑であること。もう1つは、常染色体とX染色体における突然変異率の差を十分に考慮していないということである。X染色体の突然変異率は常染色体より小さいことが知られている。体内で生成されるオスの精子の数はメスの卵子より多く、細胞分裂にともなってDNAの複製エラーすなわち突然変異が起きる機会が精子に多くなる。父からのみ子に受け継がれるY染色体は突然変異率が高くなり、母から受け継がれる割合が高いX染色体は突然変異率が低くなる。父母から均等に受け継がれる常染色体の突然変異率はそれらの間に位置する。従ってX染色体と常染色体の突然変異率の差を正しく見積もっていない場合には、X染色体と常染色体の種分岐年代を誤って推定してしまう可能性がある。
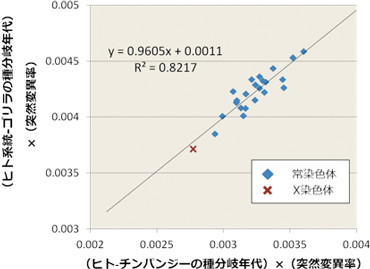
ヒトとチンパンジーの種の分岐問題に決着をつけるために網羅的ゲノムデータを用いて解析を行った。ヒト、チンパンジー、ゴリラ、オランウータンの1.9 Gb (19億塩基対) にわたるゲノムアラインメントを作成し、集団遺伝学的モデルに基づいてヒトと類人猿の祖先における種分岐年代および集団サイズを推定した。種分岐年代は、まず「種分岐年代と(1年ごとの)突然変異率の積」として推定され、後述するように適切な突然変異率を代入することにより種分岐年代自体が推定される。ヒト-チンパンジーにおける「種分岐年代と突然変異率の積」は染色体ごとにばらつき、X染色体では小さく推定された。さらに、ヒト-チンパンジーより古いヒト系統-ゴリラの種分岐についても同様に「種分岐年代と突然変異率の積」は染色体ごとにばらつくこと、またこれら別々に起こった分岐において、染色体ごとのこの値の分布は強く相関することがわかった(図4)。この相関は、「染色体間で分岐年代が異なる」か「染色体間で突然変異率が異なる」のいずれかで説明できる。
「染色体間で分岐年代が異なる」という仮説を説明するには、複雑な仮定を置く必要がある。ヒト-チンパンジーの種分岐とヒト系統-ゴリラの種分岐は時を隔てて別々に起きた出来事であるにもかかわらず、2つの種分岐年代が染色体間で相関するという現象を説明しなければならない。一方、「染色体間で突然変異率が異なる」という仮説は、染色体ごとの進化メカニズムの差異としてよりシンプルに説明できる。このことから、ヒト-チンパンジーの種分岐年代は染色体間で単一である、すなわち、ヒトとチンパンジーの種の分岐は交雑を考慮せずとも1回の種分岐として説明できることが示された。この研究結果は、パターソンらが2006年に提唱した「ヒトとチンパンジーは祖先において交雑し、その痕跡がX染色体に残る」という仮説を強く否定するものである。この解析には膨大な計算量を要した。一般的なパーソナルコンピュータでは数カ月かかる計算を、2011年に産総研に導入された大型計算機
AIST Super Cloudを用いたことにより数日で解析できた。
 |
|
図4 染色体ごとに推定した「種分岐年代と突然変異率の積」における相関
ヒト-チンパンジー種分岐およびヒト系統-ゴリラの種分岐において染色体ごとに種分岐年代と突然変異率の積を推定しプロットしたところ、これら種分岐に関する2つの値に強い相関が示された。このことは、染色体ごとの推定値のばらつきが種分岐年代ではなく突然変異率の違いによることを示している。 |
種分岐年代と突然変異率の積の推定値に適切な突然変異率を代入することにより、種分岐年代を推定することができる。近年のヒト親子ゲノム解析により明らかになった突然変異率を用いると、ヒト系統とチンパンジー、ゴリラ、オランウータンとの種分岐年代はそれぞれ600-760万年前、760-970万年前、1500-1900万年前と推定された。同様にそれらの種分岐に対応する祖先の系統における有効集団サイズ(個体数)は、59,300~75,600、 51,400~66,000、 159,000~203,000であった。これらのゲノム配列から推定された分岐年代は、これまで知られてきたヒト祖先の化石の年代とよく合致している(図1)。最古の人類と考えられるアルディピテクス・カダッパ(520-580万年前)は、ヒト-チンパンジー種分岐直後のヒト系統に属する種と考えられ、約1000万年前のアフリカに暮らしていた大型類人猿、チョローラピテクス、ナカリピテクスはヒト、チンパンジー、ゴリラの共通祖先であると示唆された。
本研究の発見から、ゲノム解析に基づく種の分岐の情報と化石記録を統合することにより、ヒトの進化の歴史を詳細に理解できるようになると期待される。さらに本研究の成果は、ヒトゲノムの産業的活用における基礎となる。本研究の発見をもとにヒトゲノムにおけるヒト特異的な性質を同定でき、これらの性質はヒト遺伝性疾患の解明およびヒトゲノムを中心とした創薬・治療法の確立に有用な情報となると期待される。
また、本研究では新たな課題も発見された。本研究の解析結果からは、染色体ごとに異なる突然変異率が同定されたが、その原因は謎のままである。ゲノムの進化メカニズムの解明は医学・生物学の広い分野に影響を及ぼす重要分野であり、さらなる研究を行っていきたい。
独立行政法人 産業技術総合研究所
バイオメディシナル情報研究センター 分子システム情報統合チーム
招聘研究員 今西 規 E-mail:t.imanishi*aist.go.jp(*を@に変更して送信下さい。)